PCR Mastermix Berechnung?
Muss den folgenden PCR-Mix berechnen, bin da komplett verloren. Wäre schön wenn jemand helfen könnte:)
Btw: Ich weiß es sollte irgendwie mit C1 * V1 = C2 * V2 gehen, komme aber trotzdem nicht drauf.
1 Antwort
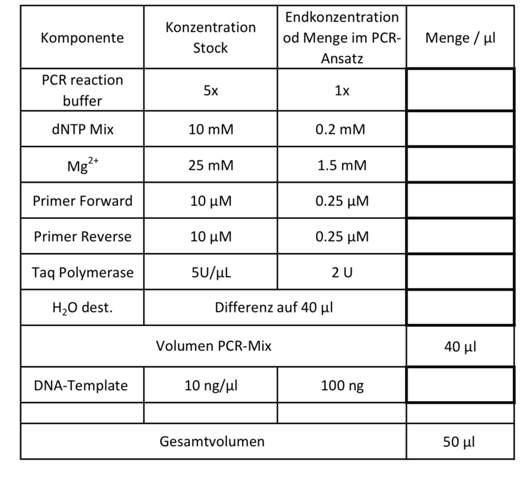
Du musst die Verdünnungsgleichung nach V1 umstellen, das ist das gesuchte Volumen, das dem Mastermix hinzugefügt werden soll. Dann stehen auf der rechten Seite drei Grössen:
- V2 ist das finale Volumen, bei dem die gewünschten Konzentrationen erreicht werden sollen. In diesem Fall sind das einfach 50 µl.
- c1 ist jeweils die Konzentration vor der Verdünnung (also das, was in der Spalte „Stock“ steht).
- c2 ist die gewünschte Konzentration nach der Verdünnung, also das in der Spalte „Endkonzentration“.
Die Einheiten solltest du immer mitschreiben. Es ist hier keinerlei Umrechnung von Einheiten nötig, die Einheiten für die Konzentration kann man „wegstreichen“.
Das 5x und 1x soll kein Grund zur Verwirrung sein. Man kann es genau mit dem gleichen Vorgehen hinkriegen, die beiden x kann man „wegstreichen“, es bleiben nur µl übrig.
Vom DNA-Template braucht es 10 µl. Das kann man als Differenz zwischen 40 µl und 50 µl berechnen oder mit der Verdünnungsgleichung, indem man sie wie beschrieben umstellt und dann den Ausdruck über dem Bruchstrich (c2 · V2) durch 100 ng ersetzt. Die Verdünnungsgleichung sagt ja aus, dass sich die Stoffmenge bzw. die Masse bei der Verdünnung nicht ändert. Hier ist nun die Masse nicht als Produkt von Konzentration und Volumen, sondern direkt als Masse gegeben, also darf man sie auch so hinschreiben.
Ähnlich ist es mit der Polymerase. Die Units (U) stehen für eine Stoffmenge. Also kann man direkt (c2 · V2) durch 2 U ersetzen.
In der Praxis würde man es aber wahrscheinlich "intuitiv" berechnen. Sowas wie "Ich will 2 U drin haben. Die Stock Solution hat 5 U pro µl. Wenn ich also 1 µl hinzufüge, habe ich 5 U drin, das sind 2.5-mal zu viel. Also muss ich 2.5-mal weniger als 1 µl hinzufügen, damit ich 2 drin habe, aka 1 µl / 2.5 = 0.4 µl." Wenn man dagegen die Verdünnungsgleichung nimmt, kommt man mit V1 = (2 U) / (5 U µl–1) = 2/5 µl = 0.4 µl hin.
Melde dich, wenn du ein Beispiel brauchst.
LG, TheGuyOfReason