Resistenzgen (ein Typ von Reportergen) z.B. Ampicillinresistenzgen?
Das ist unser Lernziel. Ich verstehe 0. Was ist überhaupt ein Resistenzgen? ist das auch Teil von der Klonierung? oder wozu braucht es das?
Herzlichen Dank für Ihre Hilfe!
1 Antwort

Ein Resistenzgen ist eine genetische Information, die dem Bakterienstamm, der es besitzt, eine Widerstandsfähigkeit gegen ein bestimmtes Antibiotikum verleiht. D.h. es kann prinzipiell auf Nährböden, die dieses Antibiotikum enthalten, wachsen.
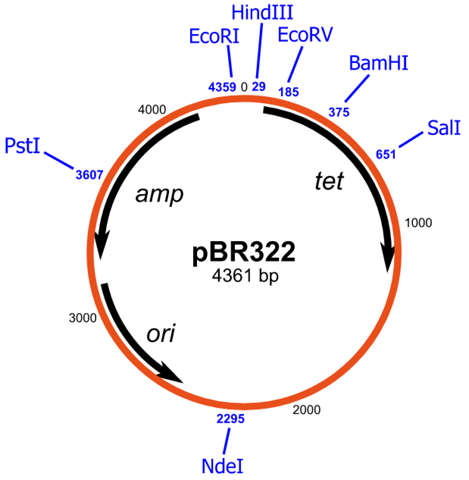
Das bringt verschiedene Vorteile bei der Klonierung. Nehmen wir mal an, der Klonierungsvektor, also das DNA-Konstrukt (Plasmid), welches die in den Organismus einzubringende DNA enthalten soll (durch Einfügen), sieht so aus wie das Plasmid pBR322:
Bildquelle: https://upload.wikimedia.org/wikipedia/commons/5/5b/PBR322.svg
Dann finden sich auf pBR322 zwei dieser Resistenzgene: "amp" und "tet" gegen die Antibiotika Ampicillin und Tetracyclin.
Diese Resistenzen sind nützlich, denn wenn dieses Plasmid als Vektor verwendet wird, um mit ihm zusammen DNA in eine Zelle einzubringen, dann bemüht man sich darum, das ganze Konstrukt der ringförmigen DNA in die Zelle zu bekommen. Denn nur so verpackt, kann die DNA in der Zelle gut überleben und ihre Information wird vermutlich abgerufen.
Es gibt jetzt mehrere denkbare Fälle über den Verlauf dieses Einbringens in Bakterienzellen:
- Bakterien ohne aufgenommenen Plasmidvektor
- Bakterien mit aufgenommenem Plasmidvektor, aber ohne eingebaute Fremd-DNA
- Bakterien mit aufgenommenem Plasmidvektor und mit eingebauter Fremd-DNA
Und diese Fälle kann man mit Hilfe der Antibiotika-Resistenzen unterscheiden bzw. überprüfen.
Für den ersten Fall hätten Bakterienzellen weder den Vektor noch ein Resistenzgen aufgenommen und wären gegen beide Antibiotika sensitiv. Sie würden auf ampicillin- und tetracyclinhaltigen Nährböden nicht wachsen. Daraus kann man schließen, dass der DNA-Transfer nicht erfolgreich war.
Für den zweiten Fall wären Bakterien gegen beide Antibiotika resistent und könnten auf Nährböden, die beide Antibiotika enthalten, wachsen. Daraus kann man schließen, dass der Transfer des Vektors erfolgreich war, aber nicht, ob dieser Fremd-DNA enthält, er könnte sozusagen "leer" aufgenommen worden sein (ohne Fremd-DNA).
Für den dritten Fall und danach sucht man, kloniert man die Fremd-DNA genau in eine der beiden Resistenzen des Plasmidvektors hinein, die dadurch zerstört wird. Das nennt man Insertionsinaktivierung. Sagen wir die Fremd-DNA soll z.B. an der BamHI-Schnittstelle mitten in die Tetracyclinresistenz eingefügt werden. Ein Bakterium, das so einen Klonierungsvektor aufgenommen hat, ist dann gegen Ampicillin resistent und gegen Tetracyclin sensitiv. Und genau an diesem Wuchsverhalten auf entsprechenden Nährböden kann man eine erfolgreichen DNA-Transfer mit Fremd-DNA dann identifizieren. D.h. die Bakterien bei denen es erfolgreich verlaufen ist, wachsen auf ampicillinhaltigem Nährboden, aber nicht auf tetratyclinhaltigem Nährboden. Daraus kann man schließen, dass sie den Klonierungsvektor aufgenommen haben und dieser auch die Fremd-DNA enthält. LG