Hilfe bei Biologieaufgabe über Selektion & Vermehrung von Fremd-DNA?
Hallo, ich habe ein kleines Problem mit diesen Aufgaben. Besonders zu Aufgabe 1 finde ich keinen richtigen Zugang. Ich erwarte hier keine perfekte Musterlösung (möchte ich auch gar nicht) , aber ich würde mich sehr freuen, wenn mir das jemand verständlich erklären könnte, sodass ich die Aufgaben lösen kann. Vielen Dank schon mal im Vorfeld !
1 Antwort
Hi,
in M1 geht es um die Aufnahme von Fremd-DNA in Bakterienzellen, einen Vorgang, den man als Transformation bezeichnet, steht auch im Text "erfolgreich transformierte Zellen auslesen". Die Auslese wird auch als Selektion bezeichnet.
Die Fremd-DNA wird dazu nicht als Stück lineare DNA in die Zellen gebracht, denn so würde sie rasch abgebaut. Sondern man verwendet ein DNA-Konstrukt, in das man die Fremd-DNA zuvor einfügt, einen sog. Vektor. Nun bringt man das gesamte Konstrukt in die Zelle. Der Vektor ist hier ein ringförmiges Plasmid "Original-Plasmid", es enthält als sog. Klonierungsvektor eine Schnittstelle zum Aufschneiden der Plasmid-DNA, BamHI und zwei Gene für Antibiotikaresistenzen gegen Ampicillin und Tetracillin, zur späteren Selektion der erfolgreich verlaufenen Transformation. Vom Aufbau her entspricht dieser Vektor dem Plasmid pBR322:
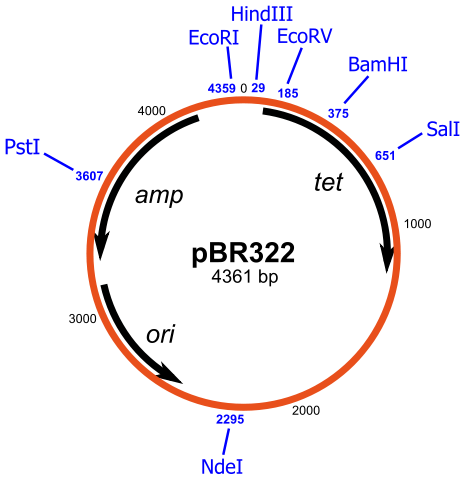
Plasmid pBR322 als Klonierungsvektor. Bildquelle: gemeinfrei link: https://commons.wikimedia.org/wiki/File:PBR322.svg
Man findet auf pBR322 alle beschrieben Merkmale wieder:
- amp: Ampicillinresistenz
- tet: Tetracyclinresistenz
- BamHI-Schnittstelle in Tetracyclinresistenz
Nun wird die Fremd-DNA in diesen Vektor eingefügt, indem man ihn zunächst öffnet. Über die BamHI-Schnittstelle wird er geöffnet und zu einem linearen Stück DNA. Schneidet man die Fremd-DNA auch mit BamHI, also dem gleichen Restriktionsenzym, dann passen die DNA-Enden von linearisierten Vektor und Fremd-DNA zueinander und bei einem erneuten Wiederverschluss des Vektors, kann theoretisch auch das Stück Fremd-DNA im Vektor eingefügt sein. Das wäre das Ziel.
Man kann den Bakterien weder ansehen, ob sie den Vektor überhaupt in sich aufgenommen haben oder falls ja, ob der Vektor, den sie aufgenommen haben, die Fremd-DNA enthält. Daher besitzt der Vektor zwei Selektionsmarker, die Antibiotikaresistenzen. Mit Hilfe derer man nun testen kann, ob der Transfer der Fremd-DNA geglückt ist.
Wir können folgende Situationen erwarten:
- Bakterien ohne aufgenommenen Vektor
- Bakterien mit aufgenommenen Vektor, aber ohne eingebaute Fremd-DNA
- Bakterien mit aufgenommene Vektor und mit eingebauter Fremd-DNA
Überträgt man ein Gemisch aus solchen Bakterien auf Nährboden mit Ampicillin (A) ergibt sich folgendes Bild: Es bilden sich diverse Bakterienkolonien aus. Kannst du so einzeichnen, wie rechst oben bei M2, zufällig verteilte, kleine runde Flecken auf dem Agar. Welche Eigenschaften haben die Bakterien dieser Kolonien? Es können keine Bakterien ohne aufgenommenen Vektor sein, da diese keine Antibiotikaresistenz besitzen und daher auf ampicillinhaltigem Nährboden nicht wachsen können. Es müssen Bakterien sein, die den Vektor erfolgreich aufgenommen haben, also Bakterienzellen mit dem Plasmid in sich drin. Jedoch hat der aufgenommene Vektor die Fremd-DNA eingebaut? Das kann man an dieser Stelle noch nicht sagen.
Dazu werden die Kolonien aus A auf tetracyclinhaltigen Nährboden übertragen (B). Dazu gibt es Stempeltechniken, wo man einfach einen mit Samtstoff überzogenen Stempel auf die Platten in A aufdrückt und dann einige Zellen der Kolonien von A, in gleichger Lagebeziehung zueinander, auf leere Platten von B "stempelt". Diese Zellen vermehren sich dort und wachsen zu gleichen Kolonien heran. Diese Methode nennt man Replikaplattierung. A wäre also die Masterplatte und B eine Replikaplatte, das heißt ein 1:1 Abdruck von der Masterplatte.
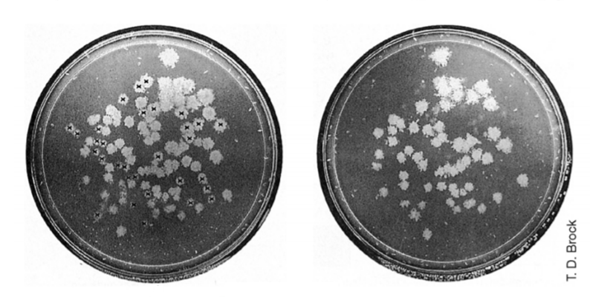
Nun zeigt sich auf der Replikaplatte folgendes Bild: Es wachsen auch dort Kolonien auf dem Nährboden. Diese stimmen alle in ihrer Lage zueinander mit der Masterplatte überein. Jedoch an manchen Stellen fehlen Kolonien, die die Masterplatte hat. Dazu sieht man hier ein Beispiel:
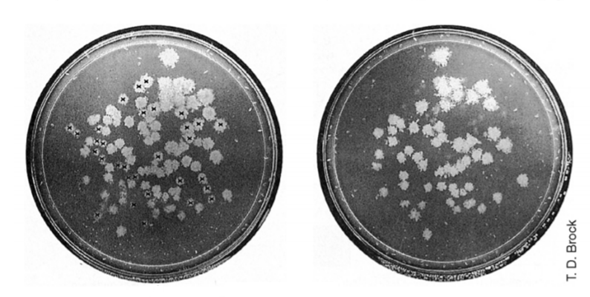
Replikaplattierung: Masterplatte (links) wie in "A" erwartet, Replikaplatte (rechts) wie "B" erwartet. Mit Kreuzen markierte Kolonien in A fehlen in B. Bildquelle: http://mikrobiologie.uni-saarland.de/lehre/2019%20Kapitel%2010,11.pdf
Was ist da los? An den Stellen wo bei B Kolonien fehlen, sind keine Bakterien gewachsen. Warum? Die Platten in B enthalten Tetracyclin, d.h. bei den Bakterien, die den Vektor aufgenommen haben und dieser auch die Fremd-DNA enthält, ist die Tetracyclinresistenz verloren gegangen, da die Schnittstelle für die Fremd-DNA genau im Resistenzgen für Tetracyclin liegt. Der Einbau der Fremd-DNA führt also zu einem Verlust der Tetracyclinresistenz und damit zu der Unfähigkeit auf Nährböden in B zu wachsen. D.h die Kolonien auf der Masterplatte (A), die dem leeren Bereich auf der Replikaplatte (B) entsprechen (die mit den Kreuzchen markiert wurden), sind Bakterienkolonien, die den Vektor mit einbgebauter Fremd- DNA haben.
Daher musst du in B auch Bakterienkolonien einzeichnen, die in Lage und Verteilung denen von A entsprechen, nur an einigen Stellen Kolonien weglassen.
Die Kolonien in A, die den weggelassenen in B entsprechen, sind erfolgreich klonierte Bakterienkolonien, mit Vektor und Fremd-DNA. Die Kolonien in A, die den in B vorhanden Kolonien entsprechen, sind Bakterienkolonien mit aufgenommenem Vektor, der keine Fremd-DNA enthält. Man würde sich zur weiteren Untersuchung also nur für die ersten interessieren, sie entnehmen und weiter verwenden.
Bei der Gensonde in M2 geht es ebenfalls um das Aufspüren von DNA-Sequenzen. Nur geht man hier anders vor und verwendet das Verfahren der Hybridisierung mit DNA-Stücken bekannter Basenfolge (komplementär zu der DNA die man sucht), die zusätzlich radioaktiv markiert ist.
Zunächst werden die zu untersuchenden Kolonien auf einer Membran ("Filter") aus Nitrocellulose fixiert, dann zusammen mit der radioaktiven Gensonde in eine Lösung gebracht. Diese Abb. vereinfacht und reduziert das Verfahren sehr stark. Jedenfalls hybridisieren dann die Sonde und die gesuchte DNA-Sequenz (falls vorhanden) miteinander, da sie komplementäre Basensequenzen aufweisen.
Anschließend wird die Nitrocellulosemembran ("Filter") "entwickelt". Hier wurde auch wieder unglaublich viel übersprungen, so dass es dir als Schüler/in natürlich schwerfällt, das zu beschreiben. Die radioaktiv markierten Sonden, die erfolgreich hybrdisiert sind, senden ja radioaktive Strahlung aus. Wenn man diese Membranen nun auf Röntgenfilm auflegt, dann wird an den Stellen, wo sich radioaktiv markierte DNA befindet, dieser Röntgenfilm durch die radioaktive Strahlung geschwärzt. Das ist mit "Filter entwickeln" gemeint. 3 Kolonien sind dort schwarz eingezeichnet bzw. sie haben den Röntgenfilm geschwärzt. Dort hat also Hybridisierung von DNA stattgefunden, demenstprechen findet sich dort die gesuchte DNA-Sequenz.
Durch Vergleich der Schwärzungen des entwickelten Filters kann man dieses Ergebnis über die ursprüngliche Petrischale mit den Bakterienkolonien legen, von denen man den Abdruck gemacht hat und die Kolonien identifizieren, die die gesuchte DNA enthalten. LG
gerne, na ja, das ist dein Unterrichtsfach. Das was ihr da machen müsst, hat diesen Hintergrund. Es ist lediglich die Frage, auf welchem Level man es verstehen will. Da das hier ja ein öffentliches Forum ist und vielfach nachgelesen wird, habe ich es umfassend erklärt, je nach Bedarf. LG
Dankeschön, auf den ersten Blick nur sehr viel Gefachsimpel aber nach ein paarmal durchdenken sehr hilfreich.