DNA Sequenzierung Guanin?
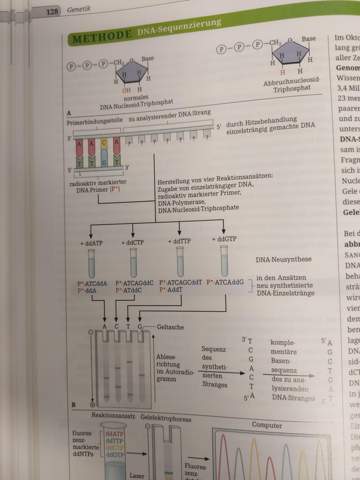
Heyy ich hab eine kurze Frage zum Abschnitt B ,wo die einzelnen Reagenzgläser gezeigt werden. Was ich mich dort Frage ist, wieso nur beim Guanin (+ddGTP) unter dem Reagenzglas nur 1 statt (wie bei allen anderen) 2 neu synthetisierte DNA-Stränge sind :/
Kann mich jemand bitte aufklären? Danke im Voraus
2 Antworten
In diesem Fall, weil der zu sequenzierende Abschnitt aus sieben Basen nur ein G enthält, aber alle anderen Basen jeweils zweimal.
Das ganze ist die Sangersequenzierung "von Hand", die älteste und zeitaufwendigste Sequenzierungsmethode. In jedem der vier Ansätze wird ein geringer Anteil eines einzelnen didesoxy(dd)-Nukleotids gegeben, der zum Abbruch der DNA-Synthese führt, wenn er eingebaut wird. Insgesamt entsteht damit für jede Basenposition ein Fragment mit einer bestimmten Länge.
Im Gel kann man dann die Fragmente der Länge nach auftrennen. Weil man vier Reaktionsansätze verwendet, braucht man auch vier Bahnen im Gel, wobei jede die Positionen einer anderen Base anzeigt. Das Gelbild zeigt dann, dass an der ersten sequenzierten Stelle an A steht, dann ein T usw.
P* steht hier für den Primer, also den bereits bekannten Sequenzbereich, den man als Ausgangspunkt für die DNA-Synthese braucht. Das TP z.B. in ddATP steht für "-triphosphat", also die dreifache Phosphatgruppe, die zum einzelnen DNA-Baustein gehört, aber für die Sequenzierung nicht von Bedeutung ist.
Der zu analysierende DNA-Strang besteht aus zwei Adenin-Basen, zwei Thymin-Basen, zwei Cytosin-Basen, aber nur einer Guanin-Base.
Demnach gibt es bei dem Guanin-Reaktionsansatz nur DNA-Stränge mit einer Länge, da das Abbruchnukleosid immer nur an der einen Guanin-Base ansetzen kann.
Bei allen anderen Reaktionsansätzen gibt es zwei unterschiedliche Ansatzstellen für das Abbruchnukleosid und daher auch DNA-Stränge mit unterschiedlicher Länge – ein kurzer und ein längerer DNA-Strang.
Somit sind mit Ausnahme von Guanin überall zwei Striche zu erkennen.
Danke für die Aufklärung! idt eigentlich zB +ddATP oder P* ddA das abbruch-nukleotid? da bin ich auch verwirrt