Genkartierung durch 3-Punkt-Analyse?
Hey, das folgende ist mein Arbeitsblatt für den Bio LK und ab Aufgabe 2 geht bei mir wirklich gar nichts mehr. Vielen Dank für jede Hilfe und Probs gehen raus an Cliff falls er das hier liest und so eine gute Antwort wie sonst auch gibt.

1 Antwort

Hi, du meinst ich habe meine Spätübungen noch nicht gemacht?
Wobei 3.) mit 1+2 nicht lösbar ist, da wichtige Informationen fehlen. Ich nehme an, dass ist Absicht, weil das ist bestimmt für die 1+ er Kandidaten, die zaubern das dann so aus dem Hut :D pass ma uff... So, dann korken wa uns ma eins auf, wa :)
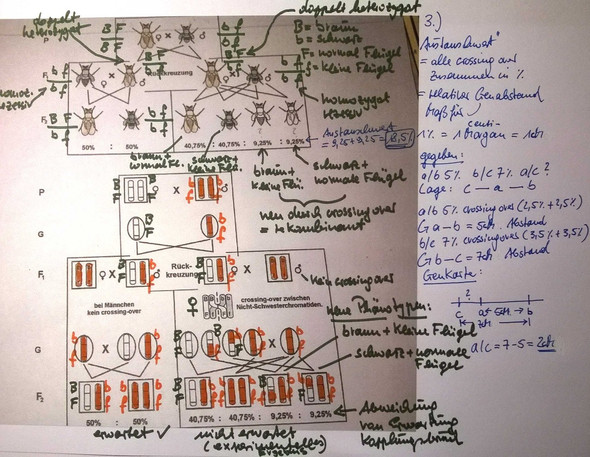
also ich hab mal eine Überleg-Skizze angehangen. Angenommen es werden homozygote Eltern gekreuzt, das ist ja unten im Genotyp so gezeichnet, dann ergeben sich die Buchstabenkombinationen, WOBEI wir Kopplungsgruppen haben, deswegen hab ich so Bruchstriche gemacht b+f sind eine Kopplungsgruppe und B+F dito.
Dann verhält sich das Ergebnis links wie erwartet 50:50 = 1:1 Mendel lässt grüßen. Rückkreuzung mit dem hier homozygot rezessiven Elter ergibt Aufspaltung 1:1 d.h. der Partner war nicht reinerbig, erwischt, was er/sie ja auch nicht ist, beide sind doppelt heterozygot, in Bezug auf beide Merkmale. Das Ergebnis entspricht also den Erwartungen.
Rechts hingegen, entspricht es nicht den Erwartungen, weil es auf einmal nur noch 40,75% und 40,75% sind, 18,5% verhalten sich anders und bilden auch neue Phänotypen, die bei Kopplung gar nicht auftauchen dürften. Diese sind nur möglich durch Crossing Over bzw. Rekombination der Allele, wobei auch die neuen Kombinationen Bbff und bbFf entstehen, woraus sich neue Phänotypen ergeben (braun+kleine Flügel; schwarz+große Flügel zu je 9,25%).
Wir haben also ein experimentelles Ergebnis, welches vom Erwartungswert abweicht und sich durch Crossing Over (Kopplungsbruch) erklären lässt.
So, das ist ja ein dihybrider Erbgang (B/b F/f), der Schritt zur Dreipunkt-Analyse wurde noch nicht gegangen, dazu muss man, wie der Name schon sagt, drei Merkmale vergleichen, vermutlich folgt das noch in Kürze... Mangels Angaben und vermutlich Vorwissen ist 3. nicht lösbar. Da ist normal das man dann ausklinkt und sagt, was ist den hier los? Blackout still running... :D
Die 1+ er Kandidaten zaubern jetzt folgendes aus dem Hut (durch Intuition, Eingebung und Recherche). Die Abweichung durch Crossing Over in % also in obigem Beispiel 9,25% + 9,25% = 18,5% Rekombinanten, nennt man den "Austauschwert". Das ist eine Maßeinheit für den Abstand der Gene auf dem Chromosom. Die Überlegung: Je weiter zwei Allele voneinander entfernt sich, desto größer ist die Wahrscheinlichkeit eines Kopplungsbruchs, auf ner langen DNA-Strecke kann einiget passieren :O desto größer ist die %-Zahl der Rekombinanten. Je näher die Genorte beieinander liegen, desto geringer ist die Wahrscheinlichkeit eines Crossing Overs, ausgerechnet auf dem kurzen Abschnitt, desto kleiner ist die %-Zahl der neuen Kombinationen (Rekombinanten). Der gesamte %-Satz der Crossing Over zusammen, ist ein Maß für den relativen Abstand zweier Gene, wobei 1%, zu Ehren des Meisters, die Einheit "Morgan" bekam (cM), hatte das mit Mg abgekürzt jedoch: https://de.wikipedia.org/wiki/Centimorgan deswegen hab ich ein c noch reingepresst :)
Daraus ergibt sich, dass a---b 5 cM voneinander entfernt liegen und b---c 7cM voneinander entfernt liegen. Wenn man nun eine kleine Skizze mit c---a---b macht und die beiden Entfernungen der Genorte einträgt, kann man, durch Subtrahieren von 7-5 den Abstand c---a errechnen, der gefragt war = Austauschwert a/c = 2% bzw. das entspricht 2 centi-Morgan. Gruß und N8!