Ph-Wert berechnen?
Kann mir vielleicht jemand sagen, ob meine Werte stimmen?

a) 3 ; b) 1.699 ; c) 1.69

a) 12 ; b) 10.7 ; c) 11.625

a) 0.6 ; b) 13.601 ; c) 3.66
2 Antworten

- a und b stimmen, c ist falsch
- a und c stimmen, b ist falsch
- a stimmt, b ist falsch, c ist unsinnig, weil sich H₂S nur zu 0.1 mol/l in Wasser löst. Unter der Annahme, eine solche Lösung wäre möglich (Druck), stimmt der pH ungefähr (mit Kₐ=1.1⋅10⁻⁷ mol/l kriege ich 3.68 heraus)
Was ist jetzt schiefgegangen?
- Essigsäure ist schwach; eine 0.1 mol/l Lösung einer starken Säure hätte pH=1, wenn Du für Essigsäure 1.69 herausbekommst, dann ist das definitiv viel zu saurer. Mit pKₐ=4.75 bekomme ich 2.88.
- Beim Kalkwasser hast Du nicht nachgedacht: Ein Mol Ca(OH)₂ liefert zwei OH⁻-Ionen, also ist bei c(Ca(OH)₂)=0.0005 mol/l die Hydroxidkonzentration c(OH⁻)=0.001 mol/l und daher der pH=11.
- Wie kann man das KOH-Beispiel mit pH=13 versemmeln?
Die Beispiele habe ich mit den Universalformeln
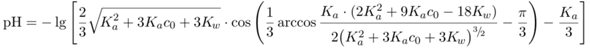
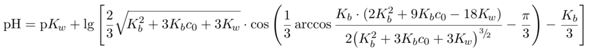
für Säuren bzw. Basen berechnet, aber eigentlich solltest Du mit einfachen Näherungsformeln dasselbe herausbekommen.
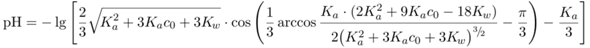
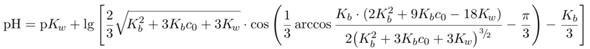

Du errätst die Phasen, fouriertransformierst das Streumuster und kriegst Deine Elektronendichte in der Elementarzelle. Elementar.
Dafür krieg ich von organischen Namensreaktionen Kopfschmerzen.

Ich schlage mich gerade mit Hakar-Ebenen herum :-(

Der Name läßt bei mir nichts klingeln.

Du kannst bestimmte Gruppen bei Proteinen mit Schwermetallionen markieren und praktisch einen Ersatz (Replacement) machen, ohne dass die Struktur darunter leidet. Da zum Beispiel Hg-Ionen deutlich größer sind als Kohlenstoffatome ist die Streuung und somit die Genauigkeit nachher höher - diese Methode nennt sich "Multiple Isomorphic Replacement". Dort, wo die Symmetrie der ersetzten Atome (Gruppen) am deutlichsten erkennbar ist (aufgrund der Anzahl der Elektronen) entstehen wie bei einer Höhenkarte deutliche Markierungen (Ringe). Vergleichst Du diese mit dem Ursprungs-Streubild kannst Du mithilfe der Patterson-Funktion (und Fourier-Transformation) auf die Distanzen zurückschließen und somit auf die Lage, Abstände und Winkel - Du löst damit das Phasenproblem.
Du tauchst praktisch Deinen Protein Kristall in verschiedene Lösung (Platin, Gold Quecksilber oder Lanthaniden-Salzlösungen ein) -> Kovalente Bindungen mit bestimmte Gruppen/Atome


Sehr gut, es sollte alles stimmen!
Universalformeln...oh je - davon bekomme ich um diese Uhrzeit Kopfschmerzen. Möchtest Du für mich nicht Röntenstrukturanalyse lernen :D