Wie erkenne ich eine Verwandtschaft bei Aminosäuresequenzen?
Ich verstehe nicht genau wie ich eine Verwandtschaft bei Aminosäuresequenzen fest stellen kann. Ich weiß, dass in diesem Beispiel Arginin- und Lysin-Vasopressin verwandt sind. Zudem gibt es eine Vereandtschaft zwischen Mesotocin und Oxytocin. Das liegt unter anderem danach, dass eine Aminosäure ausgetauscht wurde, aber bei anderen Aminosäuresequenzen sind im Vergleich auch nur eine Aminosäure ausgetauscht worden. Sie sind aber nicht verwandt. Woran liegt das und kann mir jemand das erklären?
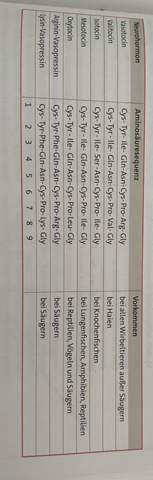
1 Antwort

Die Aminosäuresequenz wird bekanntlich bestimmt durch die DNA-Sequenz. Durch Mutation verändert sich die DNA-Sequenz im Lauf der Zeit in jeder Abstammungslinie. Unter der Voraussetzung, dass die Mutationsrate konstant ist, lässt sich das für Verwandtschaftsrekonstruktionen ausnutzen. Je näher zwei Arten miteinander verwandt sind, umso ähnlicher sind ihre DNA-Sequenzen, weil seit ihrer Trennung von ihrem gemeinsamen Vorfahren nur wenig Zeit vergangen ist, in der sich Mutationen hätten anhäufen können. Bei entfernt verwandten Arten hingegen liegt der Zeitraum ihrer Trennung von einem gemeinsamen Vorfahren länger zurück, sodass sich in jeder Linie mehr Mutationen ansammeln konnten, was zu einem größeren Unterschied in ihren DNA-Sequenzen führte.
Das gleiche Prinzip gilt damit logischerweise auch fûr die Aminosäurensequenzen, die umso unterschiedlicher sind, je verschiedener die zugrundeliegenden DNA-Sequenzen sind. Bis etwa in die 1980er Jahre hinein war der Vergleich von Aminosäuresequenzen eine weit verbreitete Methode zur Aufschlüsselung der Verwandtschaftsverhältnisse, weil die Ermittlung von Aminosäurensequenzen vergleichsweise einfach war. Erst einmal mussten Methoden der DNA-Sequenzierung entwickelt werden und als es sie endlich gab, waren sie noch sehr aufwändig und teuer. Erst mit der Entwicklung leistungsfähigerer und kostengünstigerer Methoden zur Sequenzierung der DNA wurde es üblich die DNA-Sequenzen direkt miteinander zu vergleichen. Das hat gegenüber der Vergleiche von Aminosäuresequenzen den Vorteil, dass DNA-Vergleiche genauer sind. Wegen der Degeneriertheit des genetischen Codes würden beispielsweise viele Mutationen in der Aminosäurensequenz unentdeckt bleiben. Würde z. B. durch eine Mutation aus GGG ein GGC werden, würde in beiden Fällen dieselbe Aminosäure, Glycin, eingebaut werden und die Mutation wäre damit in der Aminosäurensequenz unsichtbar.