Biologie?
Hey, brauche hier dringend die Hilfe:
bei Aufgabe 1

1 Antwort

Ein Microarray oder DNA-Chip ist eine Platte, meist aus Glas oder Kieselsäure, auf die auf einem Raster hunderte oder sogar hunderttausende winzige Punkte aus einzelsträngigen DNA-Molekülen, so genannten DNA-Sonden, aufgedruckt sind. Diese einzelsträngigen DNA-Schnipsel haben meist eine Länge von etwa 20 bp aufwärts, damit sie spezifisch genug sind, dass nur ein ganz bestimmter Genabschnitt aus einer Probe daran binden kann.
Microarrays werden häufig genutzt, um Transkriptome miteinander zu vergleichen. Als Transkriptom bezeichnet man die Gesamtheit aller mRNAs einer Zelle, also all jene Gene, die in der Zelle gerade aktiv sind (d. h. transkribiert werden). Man kann Microarrays z. B. nutzen, um die Genaktivität verschiedener Gewebesorten eines einzelnen Individuums zu ermitteln. Bekanntlich haben alle Zellen deines Körpers dieselbe Erbinformation aber etwa in deiner Leber sind vielleicht ganz andere Gene aktiv als in deinem Gehirn. Um so etwas herauszufinden, kann man Microarrays nutzen. Microarrays können auch als erster Schritt eingesetzt werden, um mögliche Gene zu finden, die eine Anpassung an die Umwelt darstellen, indem man die Genexpression verschiedener Populationen vergleicht. Wenn man z. B. das Genexpressionsmuster einer Population einer Pflanzenart einer Population von einem Standort mit mäßiger Bodenfeuchte und von einer Population an einem extrem trockenen Standort vergleicht, kann man herausfinden, welche Gene möglicherweise zu einer Anpassung an Trockenheit führen. Man kann mit Microarrays auch Vergleiche zwischen verschiedenen Arten ziehen. Mensch und Schimpanse sind beispielsweise zu 99 % genetisch identisch. Der Vergleich der Genexpressionsmuster beider Arten kann hier helfen herauszufinden, was den Menschen quasi zum Menschen macht. Und schließlich kann man sogar die Transkriptome ganzer Lebensgemeinschaften mit Microarrays vergleichen, z. B. von verschiedenen Boden- oder Luftproben.
Wie geht man dabei vor? Zunächst muss man natürlich eine Probe mit genetischem Material haben, etwa eine Gewebeprobe. Aus dieser wird die mRNA zunächst extrahiert und isoliert. Für die weitere Verwendung muss die mRNA in eine cDNA umgeschrieben werden. Das geschieht mit Hilfe des Enzyms reverse Transkriptase. Die so erzeugten cDNAs werden nun mit einem fluoreszierenden Farbstoff gekoppelt. Die Probe wird nun auf den Microarray gegeben und hybridisiert mit der einzelsträngigen DNA auf dem Chip (oder auch nicht). Beispielsweise soll die Sequenz einer einzelnen solchen DNA-Sonde (also eines einzelnen Punktes auf dem Gitter des Microarrays) "GATGCCGTACTTTCAGTATC" lauten. Wenn in der Probe ein cDNA-Fragment mit der komplementären Sequenz "CTACGGCATGAAAGTCATAG" vorhanden ist, bindet die cDNA aus der Probe an die DNA des Microarrays. Wenn in der Probe keine solche Sequenz vorhanden ist, bindet an diesem Punkt keine DNA. Nach der Hybridisierung wird der Microchip gewaschen, um alles, was nicht irgendwo komplementär an den DNA-Chip gebunden hat, zu entfernen. Nun kann man für jeden Punkt die Fluoreszenz messen. Wenn die DNA aus der Probe gebunden hat, dann ist ja an dieser Stelle auch der Farbstoff gebunden und es kann ein Fluoreszenzsignal gemessen werden, der Punkt leuchtet sozusagen. Wenn an einem einzelnen Punkt keine DNA gebunden hat, kann auch nichts fluoreszieren und der Punkt bleibt sozusagen dunkel. Anhand der Intensität des Fluoreszenzsignals kann man auch eine Aussage über quantitative Unterschiede treffen, den je mehr aktiver ein Gen ist, umso mehr cDNA ist in der Probe vorhanden und umso intensiver leuchtet der Punkt.
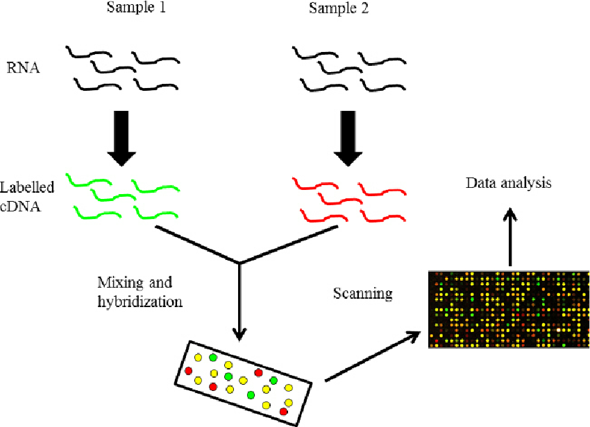
Man kann auf einen Microarray auch unterschiedliche Proben gleichzeitig auftragen, wenn man sie mit verschiedenen Farbstoffen markiert und so Unterschiede zwischen den Proben direkt sichtbar machen. Z. B. könnte man eine Probe aus der Leber mit einem grünen und eine Probe aus einem Gehirn mit einem roten Fluoreszenzfarbstoff versehen. Das sieht dann ungefähr so aus:
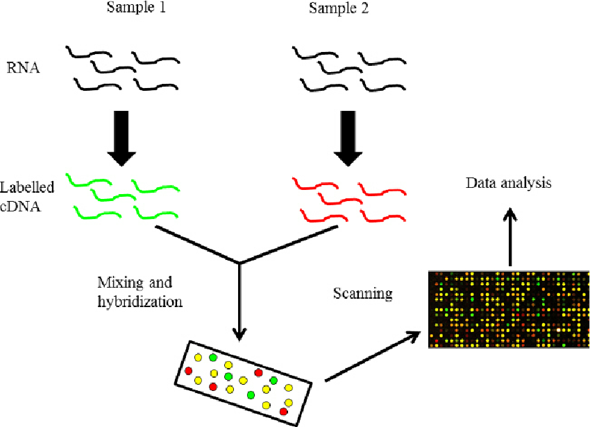
Quelle: Afzal et al. 2015.
Grün sind hier Spots, bei denen cDNA aus Probe 1 gebunden hat, bei den roten Punkten hat cDNA aus Probe 2 gebunden, bei gelben Punkten hat cDNA aus beiden Proben gleichermaßen gebunden und bei den schwarz gebliebenen Punkten hat weder aus Probe 1 noch aus Probe 2 cDNA gebunden.