Biologie - Proteinbiosynthese?
Hallo liebe Community,
Ich soll die folgende Aufgabe lösen (siehe Bild), jedoch bin ich mir nicht wirklich sicher, was genau ich hier machen soll. Sind da Fehler innerhalb der angegebenen Basensequenzen? Und wenn ja, wo sind diese und wie wirken sich diese auf die Proteinbiosynthese aus? Bin gerade wirklich am Verzweifeln und würde mich sehr über Antworten freuen!
PS: Die korrekte Basensequenz lautet
3‘ UUUCAUGGAGCCCUCAUAAGCC 5‘
Vielen Dank im voraus!


Hier fehlt eine Information, vermutlich aus Aufgabenteil (a).
Ohne die korrekte Variante können Fehler nicht identifiziert werden.

Die korrekte Variante steht irgendwo ganz anders und lautet 3‘ UUUCAUGGAGCCCUCAUAAGCC 5‘
2 Antworten

Wenn du eine Code-Sonne zur Verfügung hast, musst du schauen ob sich die Aminosäuren durch den Fehler ändern.
z.B
UUU codiert für die Aminosäure Phenylalanin(Phe).
Ändert sich der Code und man erhält UUC ist es ebenfalls Phe. Es hat also keine Auswirkungen.
Ändert sich der Code auf UUG ist es nicht mehr Phe sondern Leucin (Leu). Das bringt natürlich massive Veränderungen mit sich.
Noch drastischer wären Änderungen, die ein Stoppcodon erzeugen. Das bricht die Synthese ab und hat gravierende Folgen.
Beachte dabei aber die Richtung (5'-3'/3'-5') und überlege ob du es richtig umgeschrieben hast.

Zu 1. Das ist kein Problem. Du hast das richtig verstanden.
Zu 2. Irgendwo vor der betrachteten Region ist auch ein Startpunkt, das heißt es würde für eine Proteinsynthese schon einen Unterschied machen, aber eher für die Region zu vor. Wenn man wirklich nur den Teil hier zwischen Start und Stopp betrachten will, ist es egal was vor dem Start passiert. Also es kommt darauf an, was man betrachtet.
Wenn überhaupt kein Start vorhanden ist, könnte auch keine Synthese ablaufen. Das ist natürlich drastisch besonders dann wenn es die einzige Stelle auf der DNA ist die z.B für ein wichtiges Enzym codiert. Das kann fatale Folgen für die Zelle haben.

Die Fehler zu identifizieren, ist nun ja relativ leicht. Vergleiche die falschen Sequenzen mit der korrekten.
Dabei solltest du beachten, dass eine Mutation auch eine Insertion oder Relation sein kann, bei denen eine Base dazu kommt oder entfernt wird.
Was du anschließend machen sollst, ist wahrscheinlich lediglich die gegebenen mRNAs, in Aminosäuren übersetzen. Eine Übersetzungstabelle oder Grafik, findest du leicht online oder im Buch:
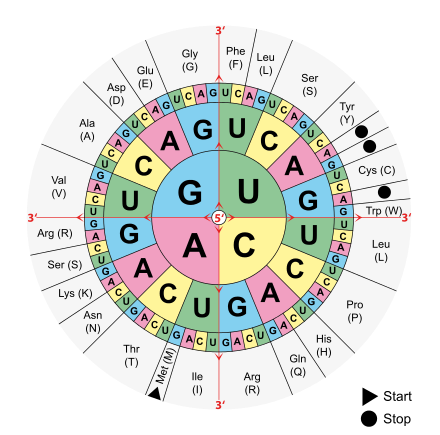
Anschließend vergleichst du die Ergebnisse für die einzelnen "Fehler", mit dem Ergebnis für die korrekte Sequenz.
Dabei kannst du davon ausgehen, dass je größer der Unterschied, in der Aminosäure-Sequenz, desto fataler die Folgenden für die Funktion des Proteins.
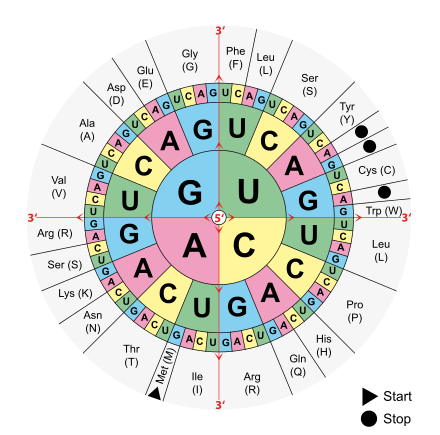
Vielen, vielen Dank für diese ausführliche Erklärung!!! Zwei kleine Fragen hätte ich noch:
Gruß