Primärstruktur auf die Basenabfolge der DNA zurückführen?
Hey,
ich schreibe morgen einen Test und muss dazu lernen wie ich die Primärstruktur der Enzyme auf die Basenabfolge der DNA zurückführe und damit das Prinzip der Proteinbiosynthese erklären.
Ich verstehe den Zusammenhang nicht und allgemein nicht wie ich das lösen kann.
Hat jemand Ahnung davon?
Dankeschön:)
1 Antwort

ist spät oder um 11 abends am Vortag
das ist die Proteinbiosynthese rückwärts, schauen wir uns das einmal an
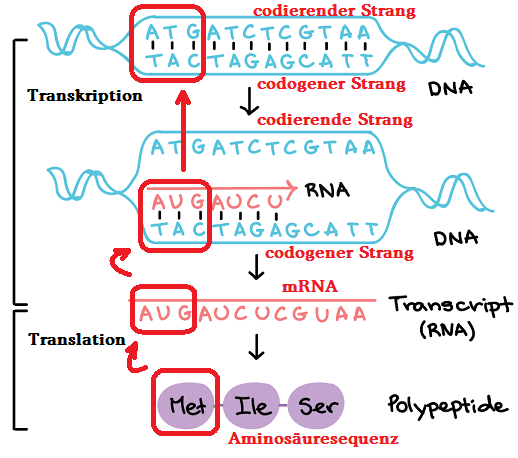
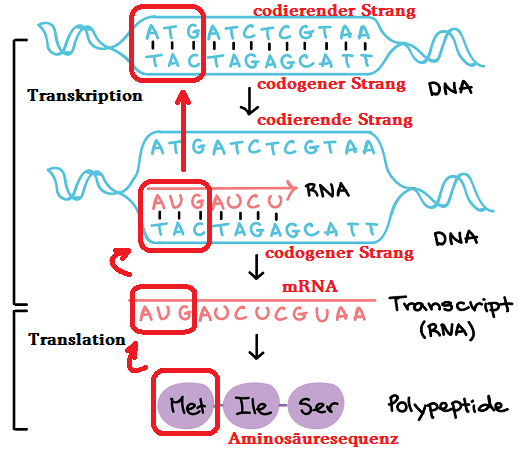
Die Primärstruktur der Enzyme (Proteine) ist die Aminosäureabfolge, z.B. Methionin(Met)-Isoleucin(Ile)-Serin(Ser)... der Polypeptidkette.
Die Information, welche Aminosäuren in der Reihenfolge zusammenzufügen sind, trägt die messengerRNA oder kurz mRNA in ihrer Basenabfolge, für "Methionin(Met)" wäre das "AUG" es sind jeweils Tripletts, also Wörter aus drei Zeichen "AUG"-"AUC"-"UCG"... man muss also die Translation rückwärts gehen.
wie man die normal (vorwärts) übersetzt steht in der Codesonne: http://www.chemgapedia.de/vsengine/vlu/vsc/de/ch/5/bc/vlus/gen_protein.vlu/Page/vsc/de/ch/5/bc/gen_protein/genet_code.vscml.html
Die mRNA erhält ihre Basenfolge als Abschrift der DNA. Da die DNA aus zwei Strängen besteht, kann man den Matrizenstrang aus der mRNA zurückübersetzen das wäre die Transkription rückwärts AUG --> TAC. Dazu muss man wissen, welche Basen miteinander paaren. In der mRNA gibt es kein Thymin(T) dafür Uracil(U). Es paaren sich in der DNA Adenin(A) mit Thymin(T), Guanin(G) mit Cytosin(C), in der mRNA statt T-->U, daher A mit U.
Das Triplett "AUG" der mRNA ergibt also als Kopiervorlage des codogenen Stranges der DNA (der, von dem die mRNA gebildet wird) A-->T, U-->A, G-->C, "TAC" als Basenfolge.
Bleibt nur noch der 2. DNA-Strang, der codierende Strang. Dieser muss dann vom codogenen Strang aus übersetzt werden "TAC": T-->A, A-->T, C-->G, "ATG" als Basenfolge. LG